單細胞轉錄組聚類後的細胞類群如何查找資料庫來定義
- 2020 年 3 月 27 日
- 筆記
- 文獻速遞(簡短介紹,擴充知識面)
- 文獻詳解(圖文並茂帶來大家系統性學習)
- R與bioconductor技巧(書籍翻譯,妙招共享)
- scRNAseq的GitHub書籍翻譯(原汁原味的名校教程)
- 全網第一個單細胞轉錄組影片教程學習筆記分享
希望大家能有所收穫!!!
你現在看到的是讀者投稿(隨機欄目)
Every cell is unique—it occupies an exclusive position in space, carries distinct errors in its copied genome and is subject to programmed and induced changes in gene expression. Yet most DNA and RNA sequencing is performed on tissue samples or cell populations, in which biological differences between cells can be obscured by averaging or mistaken for technical noise.
—Nature Method
2009年,英國劍橋大學Gurdon研究所M. Azim Surani實驗室的Tang發表了第一篇單細胞轉錄組研究的文章。隨著測序成本的不斷降低和通量的不斷提高以及數據分析流程的標準化開源化,單細胞測序已經在生物學各研究領域得到了廣泛的應用,單細胞測序的特點使得科學家們看待事物的角度發生了革命性的改變,傾向於在全局的角度發現細胞異質性,傾向於研究發現細胞分化發展的動態變化過程。像是監堂的老師,除了自己的肉眼外,又多了幾個高解析度的監控畫面,極大地推動了生物學各領域的研究。
單細胞測序技術目前主要應用於腫瘤、幹細胞、發育生物學、神經生物學、藥物研發等方面(排名不分先後)。10x Genomics商業平台進駐市場後,單細胞測開始變得越來越普遍,畢竟大家都是『弄潮兒』。通常一個樣本就可以達到好幾千的細胞,所以一次10個樣本的實驗,拿到數萬個細胞的數據實屬正常。

通常我們將心愛的樣本送給公司建庫並測序後拿到初步處理(Cellranger)後的數據,再經過各種一站式單細胞轉錄組處理工具(monocle, seurat, scater 等)分析後,終於自己辛辛苦苦(拿時間和錢砸出來的)獲得了『小可愛們』(降維分群的結果),這裡我們直接瞄一眼結果:
對這部分流程感興趣的童鞋公眾號搜索下這幾個包的名字就能找到更加詳細的說明(沒辦法,就是這麼貼心)
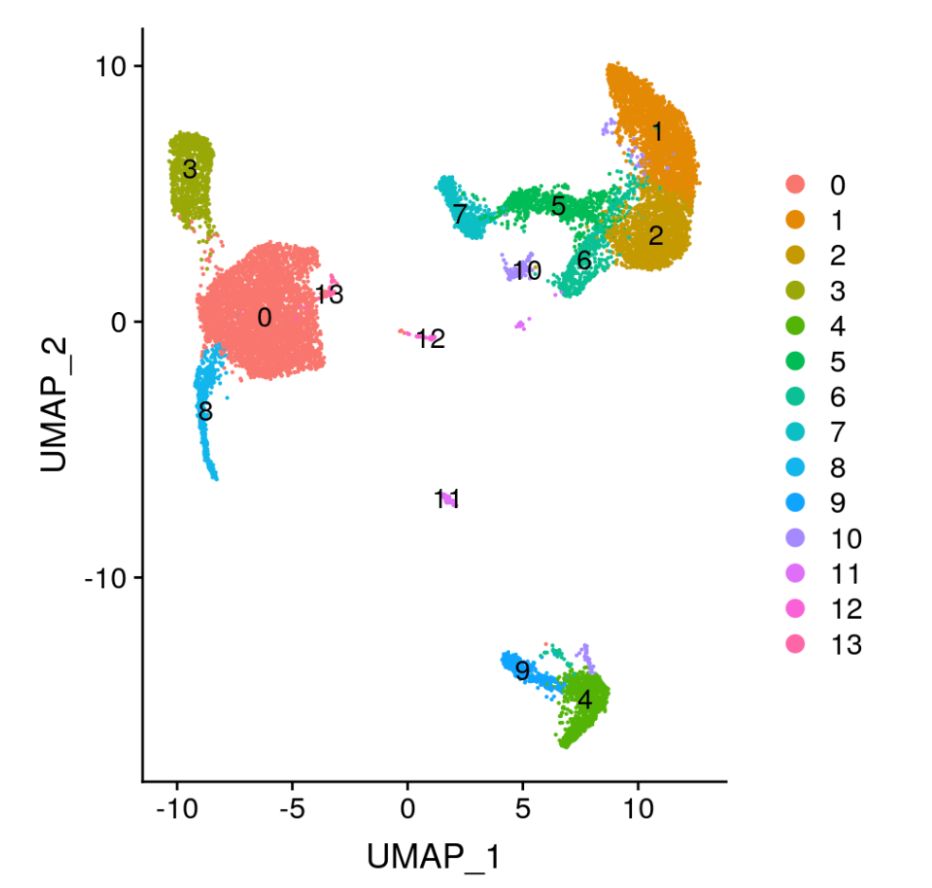
官方回答:生物學中最基本的問題之一就是哪種類型的細胞以功能協調的方式形成不同的組織和器官,單細胞轉錄組分析依據演算法得到的不同細胞群體,需要賦予其相應的生物學意義。通常是根據Marker gene來定義每一個細胞類群,可以是通過GO/KEGG資料庫進行功能富集。這樣得到的結果會比較粗糙,但對於類群不多,差異非常大的情形還是適用的。BUT上面例子的細胞類群太多,你們需要另擇他路。
幾乎每個做單細胞的小夥伴都會思考:如果有其他全面的可以參考的資料庫(必須有!別急),就再好不過了。下圖就是根據整理好的Marker gene資料庫,做出的映射,各細胞類群及其初步的比例或相互關係一目了然,是不是很棒~

看著結果一陣嘆息,跟著大佬們的教程一步步走到這裡,卻不知道,所謂的生物學意義究竟在哪兒!!!細胞類群的確定便是揭示細胞間特徵,並進行後續深入生物學問題研究的首要任務,然而面對每種細胞類群成百上千個的Marker genes,你是否會感到彷徨…
七步之內必有福利,以下細胞Marker資料庫供君參考:
CellMarker資料庫
這是哈爾濱醫科大學 Yun Xiao老師等在2019年1月份發表於核酸研究 (Nucleic Acids Research)資料庫專刊的工作,訪問地址: http://biocc.hrbmu.edu.cn/CellMarker
該團隊通過梳理100,000+發表的文獻,梳理出人的158個組織 (亞組織)的467個細胞類型的13,605個Marker基因,和鼠的81個組織 (亞組織)的389個細胞類型的9, 148個Marker基因。
- 主頁提供人和鼠的全局視圖:1. 通過單擊嵌入在網路影像中的超鏈接「人體或小鼠細胞的解剖位置」,快速探索細胞標記; 2. 單擊右上角的圖標可以切換至小鼠模式; 3. 通過人和小鼠的解剖位置,可以方便快速瀏覽所列細胞類型的細胞標記,單擊組織圖標可以顯示更詳細的細胞類型;4. 單擊細胞類型時,它將跳轉到與細胞類型相關的細胞標記基因的相應搜索結果。


- 瀏覽介面也很簡潔,頁面呈現細胞和組織的分層分類,包括人和小鼠兩個物種,從組織到細胞類型,邏輯很清晰。 1. 用戶可以通過單擊人(或鼠)的不同組織中的細胞類型來瀏覽細胞標記基因,並且可以返回匹配的細胞標記條目的完整列表。 例如,要瀏覽與人體脂肪組織相關的條目,您可以:2. 單擊「人體」,選擇「脂肪組織」;3. 找到感興趣的細胞類型,例如「脂肪來源的幹細胞」;4. 相關的細胞標記將顯示在右側面板上,包括細胞類型的細胞標記和來自不同來源的條目的統計圖。檢索到的標誌物,標誌物的醒目程度反映了文獻支援數的多少(好喜歡這個細節)!

- 另外,在結果部分還展示了這些cell type的來源(單細胞測序、實驗、綜述、公司等),非常方便大家尋找相應的來源並取捨,CellMarker不只是單細胞數據明確細胞類型聯繫生物學意義的時候可以用,有些內容對於濕實驗的小夥伴來說更具有參考價值。除此之外,用戶還可以:1. 通過輸入基因名稱,基因ID或蛋白質名稱來搜索任何感興趣的基因,以查詢特定基因可以作為細胞標記的哪些組織的細胞類型;2~3. 點擊「提交」按鈕後,搜索引擎將返回一個互動式氣泡圖和一個顯示細胞標記綜合資訊的表格。 互動式氣泡圖顯示了感興趣的基因在不同組織的不同細胞中用作細胞標記的頻率

- Details點進去則是相關基因的資訊,下方還鏈接了相關的支援文獻:

- 感興趣的小夥伴們可以登錄網站先行體驗一下,幫助里還有更詳細的的小Tips!
Mouse Cell Atlas
- 奉上重量級文章《Mapping the Mouse Cell Atlas by Microwell-Seq》鏈接:https://www.ncbi.nlm.nih.gov/pubmed/29474909

- Mouse Cell Atlas通過對Microwell-seq對40多種組織器官和40W+的單細胞進行了測序並分析得到各組織器官的細胞分類及其相應的細胞類型,同樣這裡也提供了方便讀者的網頁可視化搜索(http://bis.zju.edu.cn/MCA/gallery.html),用戶可以根據器官和基因名在資料庫中搜索得到自己想要的結果。例如我們通過分析測序數據得到了一系列的細胞類型,往往這些細胞類型被分成了諸多亞型,此時則可在特定組織器官的資料庫下對比相應的marker gene來確定自己的細胞類型並對其進行歸一或細分。下圖顯示的是小鼠的不同組織器官,用戶可根據自己感興趣的方向點進去就會顯示該組織單細胞測序的tSNE圖及其相應的分群和細胞類型marker。這個資料庫最大的優點就是所涵蓋的小鼠組織器官特別全面(雖然沒有涵蓋所有的研究領域),相信絕大部分做小鼠的小夥伴都不會錯過


- 為了滿足大家的求知慾,減輕伺服器的負擔,scMCA還在github放了R包https://github.com/ggjlab/scMCA,感興趣的小夥伴可以自行安裝體驗一下:


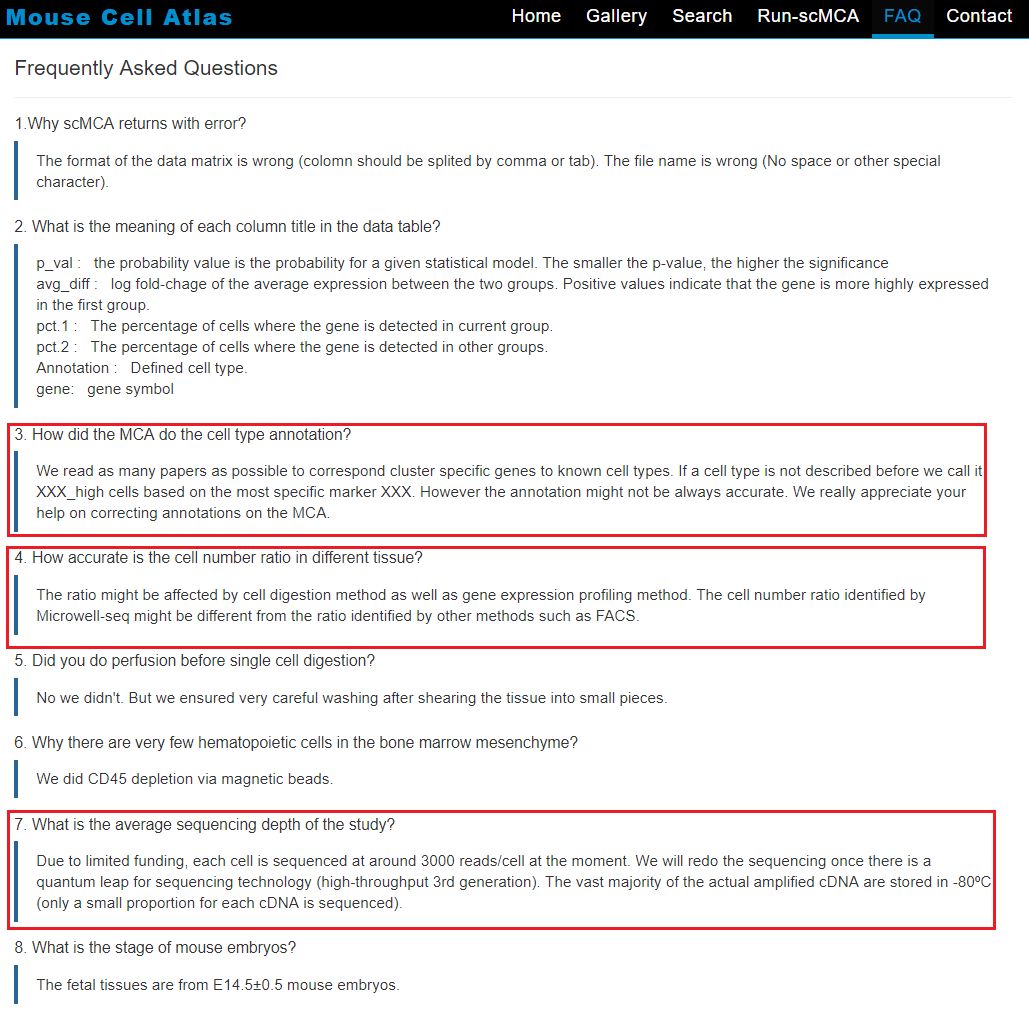
- 可能會有小夥伴們發現自己的單細胞數據分析結果和資料庫中的Marker會有偏差,不要驚慌!小編有個習慣就是在使用一個資料庫的時候先看一下背景介紹和幫助,非常有助於大家正確地學習使用資料庫,拿此資料庫為例,大家仔細看一下資料庫的背景就可以發現資料庫並不是完美的(成本太高啊!),建庫方法和分析流程也會產生一定得誤差。但是,對於研究小鼠的(特別是發育研究)客官們來說,這絕對是目前最好用的小鼠單細胞轉錄組資料庫之一了!
- 八卦放在最後,郭國驥教授在中國原創音樂基地有很多原創歌曲,底下評論說,是被科研耽誤的原創歌手哦。https://www.sohu.com/a/224004856_503422
PanglaoDB
- PanglaoDB的定位是對小鼠和人類scRNA-seq感興趣的科學界的資料庫(https://panglaodb.se),該資料庫收集並整合來自多個研究的數據(來自小鼠的170種組織954個樣本近400W細胞和來自人的68種組織279個樣本100w+細胞),並通過統一的框架呈現它們。例如在搜索細胞類型Markers的時候,用戶可以根據細胞或組織類型來進行的篩選,選定後就可以呈現出相應的Marker了

- 當搜索單個基因時,可根據資料庫主頁的介紹和使用(都寫得很清晰),點擊search就可以愉快地探索了:

signatureDB
來源於文章:Genetics and Pathogenesis of Diffuse Large B-Cell Lymphoma. 發表2018新英格蘭雜誌。
https://www.ncbi.nlm.nih.gov/pubmed/29641966
數據以表格的形式進行展示,鏈接放在下面,大家可以自行下載研究:
- SignatureDB (Excel)
- SignatureDB annotation (Excel)

最後在此提醒大家最好是在詳細了解明確各大資料庫的優缺點,明確所檢索到的Marker的相關文獻及研究背景後(例如:轉錄組層面的Marker在表達量和時間空間上並不一定完全和蛋白質組學對應;參考文獻是否可靠等),再結合自己的課題斟酌著對自己的『小可愛們『下定義,資料庫只是作為一種參考。隨著測序技術的持續發展和成本的不斷降低,多組學的合併研究(轉錄組、蛋白質組、表觀組等)和單分子成像技術(MERFISH、SeqFISH、RNAScope和DNA FISH等)的推廣,相信各大資料庫們會越來越完善。關於各資料庫的其他細節就不再劇透了,大家自行探索研究吧!

