人類結直腸癌單細胞多組學分析
- 2020 年 3 月 27 日
- 筆記
每個人的時間精力有限,必須優先閱讀相關文獻,開設這個欄目也是希望為大家推薦高質量的單細胞相關文獻。如果大家對單細胞轉錄組感興趣可以關注一下,哪怕每天只學一點點,積土成山,積水成淵。
當然一個人的力量終歸是小的,我也希望匯聚一群人,形成一個場,這裡頭最重要的生產力不是單個人多聰明,多厲害,而是每個人相互作用,形成的那個氛圍。
希望大家能有所收穫!

文章信息
文章由北醫三院付衛團隊、喬傑團隊和北大生命科學學院湯富酬團隊於2018年11月合作發表在Science上,文章題目是:Single-cell multiomics sequencing and analyses of human colorectal cancer 這一篇理解難度稍大,當做背景知識的了解,至於其中數據的分析細節文中沒有提及

1
導語
之前一次只能研究單細胞層面的基因組、轉錄組或DNA甲基化組其中一種,不能在一個細胞中同時研究多個組學,2016年湯富酬研究組將三重組學研究方法scTrio-seq(single-cell triple omics sequencing technique)發表在Cell Research上。2018年11月北醫三院付衛團隊、喬傑團隊和北大生命科學學院湯富酬團隊又在Science合作發表了Single-cell multiomics sequencing and analyses of human colorectal cancer
文章優化了單細胞多組學測序技術,並對原發瘤、淋巴轉移和遠端轉移區域分別採樣,首次從單細胞分辨率解釋了結直腸癌的發生與轉移過程中中基因組拷貝數變異、DNA甲基化異常及基因表達改變的特點,證明了用單細胞多組測序重建遺傳譜系和追蹤其表觀基因組和基因表達動力學的可行性
2
背景知識
- 淋巴轉移(lymphatic metastases)和遠端轉移(distant metastases) 癌症可以以2種方式出現在淋巴結中:一種是從淋巴結形成的腫瘤叫淋巴瘤,另一種是從其他部位擴散叫淋巴轉移(更為常見)。淋巴轉移一般會和乳腺癌、前列腺癌、肺癌、結直腸癌的不良預後相關,淋巴轉移雖然不是致死因素,但會導致癌細胞擴散到重要器官。遠端轉移也叫惡性轉移,腫瘤細胞從原始發生的部位藉由侵入循環系統,轉移到身體其他部位繼續生長,幾乎不可能使用外科手術切除根治,多半只能用大範圍循環全身的放射治療或化療等手段來抑制已轉移的癌細胞。 2017年發表在Science的Origins of lymphatic and distant metastases in human colorectal cancer 中描述了結直腸癌之前的腫瘤擴散的TNM層級是:primary tumor(T)=》lymph node system(N)=》distant metastases(M),但是有臨床證據表明移除淋巴結並不會提高病人存活率,因此N和M之間的關係可能並不是簡單的上下級。文章發現淋巴結和遠端器官存在獨立起源的證據,只是淋巴結轉移機制形成的更快,因此淋巴轉移形成更早,發生更頻繁。因此作者不推薦直接假設淋巴結會引發遠端轉移而直接切除(https://www.medscape.com/viewarticle/882502)
- 結直腸癌colorectal cancer(CRC) :是結腸癌和直腸癌的統稱,是消化道惡性腫瘤之一。2018年Cancer Stats統計顯示:結直腸癌在男性中發病率第2死亡率第3,女性發病率第4,死亡率第3。 約95%的結直腸癌是由結腸和直腸內壁的腺細胞發展而來,癌症通常開始於內壁最內層,並緩慢生長到外層。(http://www.cancer.org/Cancer/ColonandRectumCancer/DetailedGuide/colorectal-cancer-what-is-colorectal-cancer) 結直腸癌發生率在40歲開始增加,60~75歲達高峰,結腸癌在女性患者較常見;直腸癌在男性患者常見;大約5%的結腸癌或直腸癌患者在結腸和直腸有兩個或更多病灶,並非簡單從一個病灶轉移至另一個所致(https://www.msdmanuals.com/) 結直腸癌典型的分子特徵是:基因組不穩定性、表觀遺傳學異常、基因表達紊亂
- 瘤內異質性 Intratumoral heterogeneity (ITH) :惡性腫瘤的特徵之一,腫瘤異質性包括腫瘤間異質性(不同腫瘤細胞之間的基因與表型不同)和腫瘤內異質性(相同腫瘤細胞以內的基因與表型也不同),其中腫瘤內異質性又包括空間異質性(相同腫瘤不同區域不同,如未擴增的細胞背景中有成簇擴增細胞;少量擴增背景中有未擴增的細胞;孤立的細胞擴增【利用多位點取樣方案或者tissue microarrays (TMAs) 調查】)與時間異質性(原初腫瘤與次生腫瘤不同)。 異質性的產生是因為同一腫瘤由多種不同基因組特徵的細胞組成,每一種細胞構成一個亞克隆(subclone)。腫瘤組織會存在對治療藥物有抗性的亞克隆,但比例不高。當治療的藥物除去敏感的亞克隆時,抗性的亞克隆細胞會不受藥物抑制並且少了空間競爭,因此會加快生長速度,導致腫瘤複發或者發生轉移,而且轉移後的亞克隆對同種治療方案也會有抗性。因此研究腫瘤細胞的亞克隆以及不同的亞克隆的轉移是一個熱點,尤其是亞克隆是如何從原位癌轉移到其他臟器而形成轉移癌。2017的一篇文章專門研究了轉移癌亞克隆與原位癌亞克隆的進化關係 ,他們發現結直腸癌腫瘤轉移癌高深度測序就可以找到腫瘤的大部分基因組變異,另外驗證了"轉移癌多克隆起源說(轉移癌是由多個起源於原位癌的亞克隆發展而來,而非由單個細胞發育)",發現了結直腸癌的淋巴與遠端並行轉移 。
3
方法
- scTrio-seq2技術:用於somatic copy number alterations (SCNAs)拷貝數變異、DNA甲基化特徵、細胞連續的轉錄信息;整合了單細胞重亞硫酸鹽測序(scBS-seq)用於全基因組甲基化分析;研究的細胞數量從之前的25個增至1900個
- 分析了12個CRC患者(III期或IV期)的約1900個單細胞,7.6Tb高質量測序數據。DNA甲基化研究中平均每個細胞測序量為4.1Gb,平均覆蓋到全基因組內870多萬CpG位點;轉錄組研究中每個細胞測序量為0.9Gb,平均覆蓋3700多個基因
- 多區域採集了10個患者的原發瘤、淋巴轉移瘤或遠端轉移瘤樣本(利用兩種不同來源的細胞,可以發現每個患者因突變而產生的遺傳譜系)
- 文章實驗流程 圖A是取樣:治療前後的腫瘤區域(包括原發瘤primary tumor,PT;淋巴結轉移位 lymph node metastasis, LN;肝轉移位 liver metastasis, ML;化療後肝轉移位 posttreatment liver metastasis, MP),然後測序分析了基因組、轉錄組、甲基化組; 圖B是化療6個周期後的患者CRC01取樣:一共取了ML(4個)、MP(5個)、LN(3個)、PT(4個)共16個腫瘤區域

- 單細胞甲基化數據處理 首先raw reads去接頭、引物、低質量鹼基,然後利用
Bismark(V0.7.6)clean reads比對到hg19基因組,PCR重複利用samtools rmdup(V 0.1.18)去除,數據統計(比對數、比對率、CpG位點數、亞硫酸氫鹽轉化率等)[其中亞硫酸氫鹽轉化率是由非甲基化的lamda DNA的spike-in計算的],CpG位點小於200萬個或亞硫酸氫鹽轉化率小於98.5%的細胞被排除。僅使用甲基化水平大於0.9或小於0.1的CpG位點進行總體甲基化水平計算。 啟動子區設定為轉錄起始位點的上游1 kb到下游0.5 kb。為了計算RefSeq基因各基因體的DNA甲基化水平,將每個基因體劃分為100個等分,並將其上下游側翼區域(15 kb)分別劃分為10個等分。基因組注釋信息從UCSC獲取。利用bedtools(V 2.17.0)和自定義腳本(沒放Git鏈接)計算平均甲基化水平,設置滑動窗口大於等於3個CpG位點 - 根據甲基化測序數據估計拷貝變異數 主要基於Garvin等人開發的Ginkgo算法 ,基因組被分成10856個不等長的bins,長度中位數為250kb,並根據算法的過濾器排除了一些異常的bins。 BED文件是利用bedtools從BAM文件得到。每個bin的read counts值利用所有bins的count平均值進行標準化,採用低水平均一化(Lowess normalization)來校正基因組GC含量的偏差。另外,以正常的二倍體細胞作為對照,減少scRS-seq的其他誤差。利用Circular binary segmentation (CBS) 對copy number文件進行分隔,參數為"
alpha = 0.0001"和"undo.prune = 0.05" 。CBS分隔後,每一段的所有bins的計數重置為這一段的bin count的中位數。每個單細胞的基本拷貝數由smallest sum-of- squares (SoS) error和 scaled copy-number profile (SCNP)決定,其中SCNP又進一步四捨五入,最後得到了整數值copy-number profile (FCNP)。利用 GISTIC2.0 (https://software.broadinstitute.org/cancer/cga/gistic) 鑒定了重要的SCNAs和潛在的基因靶點。 - TCGA數據分析 從https://tcga-data.nci.nih.gov/docs/publications/coadread_2012/獲得已發表的人類CRC的SCNA片段數據,用於SCNA頻率統計(不包括X染色體)。將本文的數據與TCGA的進行比較時,本文研究的CMS3類型的患者CRC02是找不到對應的,只有Affymetrix SNP 6.0芯片得到的178個non-hypermutated樣本。SCNA譜進一步轉變成長度不等的bins用於單細胞SCNA統計,拷貝數為>2.5的bin表示擴增,小於1.5表示缺失。Circos圖是根據https://github.com/venyao/shinyCircos製作的。
- WGS數據處理 raw reads =》trimmed =》BWA mem(V 0.7.12) 比對到hg19 =》 samtools sort =》Picard(V1.139) merge BAM文件 + 標記重複 =》BAM文件用GATK(V 3.4-46) 預處理=》muTect (V 1.1.4) call SNVs ,自己腳本過濾=》取每個患者的外周血或鄰近正常組織作為對照(somatic variants),在線Venn圖做出SNV數量
- 單細胞RNA-seq處理 利用湯教授自己的方法得到的數據處理:預處理過程都一樣,然後用STAR(V 2.5.0) 2步比對到hg19,Cufflinks(V 2.2.1) 使用默認參數進行FPKM定量; 利用multiplexed scRNA-seq方法得到的數據,先利用read2的barcode信息將reads分配到每一個細胞,每個細胞中read2對應的read1利用read ID分隔,read1中的TSO序列 利用自己的腳本過濾掉,然後利用Tophat(V2.1.1) 單端比對到hg19,利用UMI實現TPM標準化(文中說道:大部分的表達量都使用
log2(FPKM + 1)或者log2(TPM/10 + 1)) ,然後統計了mapped read numbers, mapping ratios, RefSeq gene numbers等信息,根據FPKM > 1 or TPM > 1去除了比對率 < 20%或有表達量的Refseq基因數量 < 1500
4
結果
根據甲基化數據得到的基因組拷貝數變異來推斷基因譜系
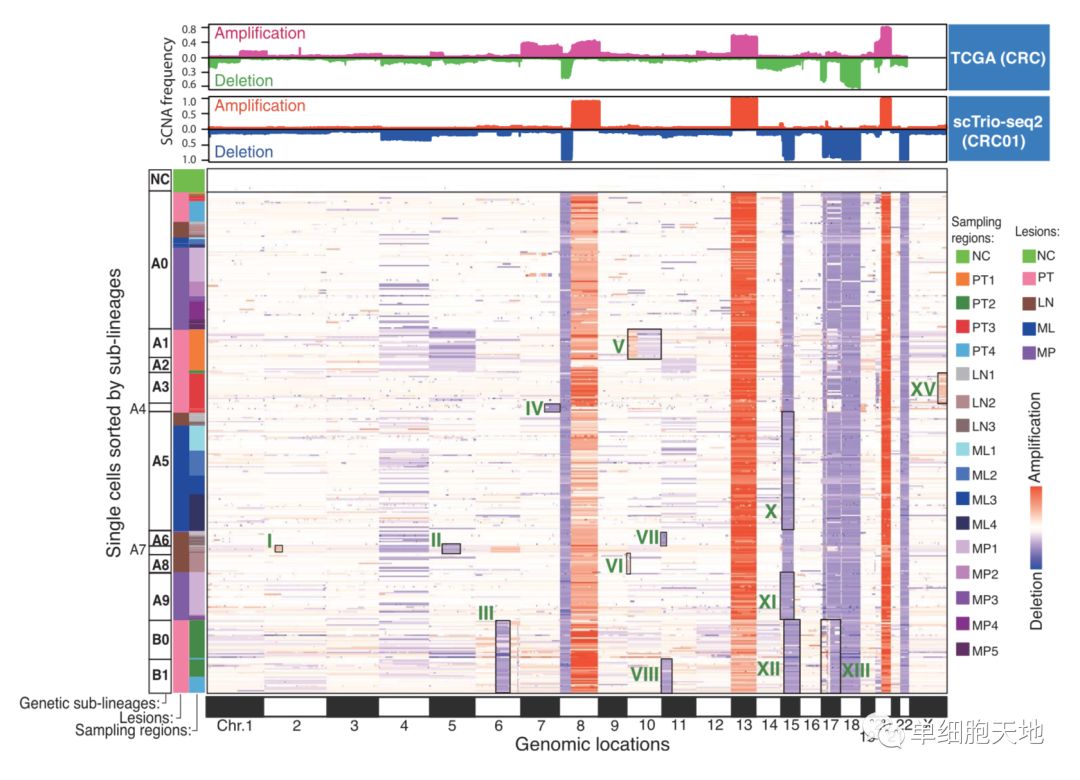
Genomic alterations in tumors provide markers for lineage tracing. 克隆變異出現在腫瘤早期階段,亞克隆拷貝數變異標誌着亞型的出現 結直腸癌患者單個癌細胞的染色體拷貝數變異譜+高精度的染色體內斷點信息=》譜系追蹤=》原發位腫瘤(PT)的亞克隆結構通常比轉移位腫瘤更複雜
結果得到了5個患者的90個細胞以上的甲基化數據,細胞被分成了不同的基因亞型
其中,CRC01基於21個亞克隆的拷貝數斷點,鑒定了來自2個不同譜系(A、B)的12個亞型,其中每個亞型都有4-8個亞克隆的斷點(斷點的上下位置和拷貝數變異數增加、減少對應),A5亞型同時出現在了肝轉移位和淋巴轉移位,表明這兩種移位有共同起源,這5個病人的原癌亞克隆結構比其他轉移類型更複雜
CRC01患者癌細胞的單細胞染色體拷貝數變異譜:
CRC01患者亞克隆拷貝數斷點:

CRC01患者亞克隆結構:

甲基化異質性
結腸直癌細胞的DNA甲基化水平要低於癌旁的正常上皮細胞,同一腫瘤組織中同一譜系的甲基化程度相近,不同譜系出現差別。低甲基化基因組區域顯著富集在LTR(long terminal repeats)、LINE-1 (long interspersed nuclear elemnt 1)和異染色質區域(H3K9me3),而高甲基化的基因組區域顯著富集在CpG島、H3K4me3和開放染色質區域。
以CRC01患者為例:

甲基化的異質性主要來自同一個患者腫瘤內不同亞克隆之間的DNA甲基化差異,而不是同一個亞克隆內部不同細胞間的DNA甲基化差異
甲基化譜和基因表達譜的相互關係
啟動子區域的甲基化與相應基因的表達呈顯著的負相關,而基因區的甲基化與相應基因的表達呈正相關

【The gray lines represents individual cells. The blue line represents the mean value for each patient. TSS, transcription start site; TES, transcription end site.】
同一遺傳譜系的腫瘤細胞在轉移過程中DNA甲基化及基因表達的變化情況
同一患者同一譜系的腫瘤細胞從原發灶到轉移灶全基因組DNA甲基化水平基本穩定,組內局部區域可能會有比較大的波動

結直腸癌去甲基化特點
每個亞型內的去甲基化 程度是一致的,不同亞型程度不同 ;
正常上皮細胞的基因組區域甲基化越高,它就越容易發生去甲基化;
去甲基化的程度與基因組重複序列L1(long interspersed nuclear elemnt 1)以及癌旁正常組織中H3K9me3修飾的密度呈正相關,與H3K4me3標記和正常組織的開放染色質區域密度呈負相關;
有趣的是,L1比LINE-2更活躍,在所有病人的癌細胞中顯示了更強的去甲基化能力,這個與胚胎髮育中情況相反(胚胎髮育過程中L1一般比L2去甲基化能力弱) ,說明在腫瘤發生與發展過程中,L1和異染色質區域產生了異常的去甲基化過程,打破了正常的發育規律
癌細胞相比於癌旁細胞的DNA甲基化水平:

染色體水平的DNA甲基化異常和基因組不穩定性
結直腸癌細胞中6條染色體(4號、5號、8號、13號、18號、和X染色體)傾向於發生更強烈的DNA去甲基化,其中三條低甲基化染色體(8、13和18)在TGCA和研究的患者中都有較高的拷貝數變異。結合WGS結果發現,有5條第甲基化的染色體(4號、5號、8號、13號、和X染色體)的單核苷酸變異(SNVs)發生顯著富集



